Normas ABNT 2024 atualizadas de acordo com as as novas regras e normas – Tudo que precisa saber e aprender. (Artigo Atualizado)

Se você está fazendo um curso técnico ou se está fazendo a sua graduação sabe que no momento certo você terá que fazer um TCC.
Quem sabe aquela temida monografia, e para seu trabalho sair conforme solicitado é importante que o estudante saiba as regras da ABNT, ou normas ABNT como deseje chamar.
Então confira algumas informações básicas sobre as normas da ABNT.
ABNT – Associação Brasileira de Normas Técnicas
A Associação Brasileira de Normas Técnicas pode ser chamada apenas de ABNT, esse é um órgão que tem como finalidade de normalização e desenvolvimento técnico. Sua fundação aconteceu em 1940.
Desde seu surgimento a sua finalidade é para contribuir com o desenvolvimento do trabalho em caráter científico e tecnológico, ou seja, sua missão é padronizar as técnicas.
E, as normas mais procuradas pelos estudantes, professores e professoras são:
- Norma 14724 – elaboração de artigos científicos, trabalhos acadêmicos (dissertação, teses, trabalho de conclusão de curso) (Baixar ABNT NBR 14724 em PDF Gratuitamente!)
- Norma 6023 – elaboração de referências
- Norma 10520 (atualizada em julho de 2023) – elaboração de citações em documentos
Estrutura do Trabalho com ABNT
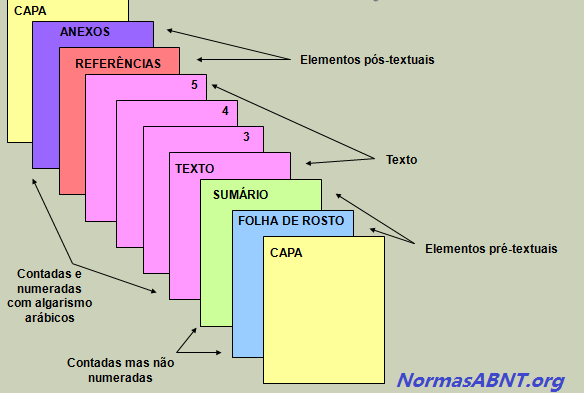
A estrutura é feita mediante as regras estabelecidas pela ABNT, sendo que essas são compostas por textuais, pré-textuais e pós-textuais, esses elementos pode ser encontrados da seguinte maneira:
Pré-textuais

É toda introdução feita no trabalho ou na monografia dando uma aparência antes do conteúdo do trabalham, esse deverá ser composto por: Capa, Folha de Rosto, Folha de Aprovação, Dedicatória, Agradecimentos, Epígrafe, Língua estrangeira, Sumário.
- A capa é o primeiro elemento identificador do trabalho científico. Deve conter os dados identificadores da instituição de ensino, o título do trabalho e o local e o ano de produção. Não é numerada e nem conta para a numeração das páginas da monografia. Deve obedecer a cor determinada pela Coordenação de Monografias para aquele ano letivo.
- A folha de rosto é o segundo elemento identificador do trabalho científico e contém alguns elementos da Capa, acrescidos de uma breve descrição do objetivo do trabalho.
- O cabeçalho pode ser utilizado em substituição à capa e folha de rosto, e NUNCA junto deles. Os itens devem ser apresentados em espaço simples e fonte no tamanho 12.
Textuais

Nesse caso, deve conter o conteúdo central do que esta sendo abordado no trabalho, sendo assim composto por: Introdução, Desenvolvimento e Conclusão. É importante frisar que é nessa etapa que o autor/escritor deverá aborda a sua problemática sugerida como trabalho, colhendo as informações por vias acadêmicas e complementares.
- Introdução
Lendo a introdução, o leitor deve sentir-se esclarecido a respeito do teor da problematização do tema do trabalho, assim como a respeito da natureza do raciocínio a ser desenvolvido.
- Desenvolvimento
Perfil ideal para o tema do trabalho. Ambiente ideal de trabalho. Objetivo Geral, Métodos.
O desenvolvimento do conteúdo obedece à organização em introdução, desenvolvimento e conclusão, observando requisitos como a gradualidade, a coesão, a coerência e a unidade.
- Conclusão
Ponto de vista dos autores sobre o tema e a conclusão do trabalho realizado.
Pós-textuais

Após o primeiro e o segundo passo dado, segue nesse momento deve conter o que vem após o conteúdo, ou seja, quais foram as formas de obter a referência para o conteúdo. Esse deve ser composto por: Referências Bibliográficas, Anexos, Índices podem ser integrados ao trabalho.
- As referências bibliográficas são colocadas depois da conclusão e apresentam a relação dos documentos consultados para a elaboração do trabalho científico. São listadas as obras citadas no corpo do texto e obras consultadas como apoio.
Tipos dos trabalhos científicos
Tanto nos cursos de graduação, quanto de Pós-Graduação utilizam-se vários tipos de trabalhos científicos. Ele podem ser classificados em três tipos conforme o seu porte, ou seja, conforme o número de páginas: em trabalhos de pequeno, médio e grande porte.
- Os trabalhos de pequeno porte, como fichamentos (resenhas, resumos e esquemas) e produções textuais mais informais (como reações, avaliações, análises e comentários) possuem de 1 a 10 páginas.
- Os trabalhos de médio porte, como o artigo científico, o projeto (projeto de pesquisa de campo; projeto de ação social e ministerial; projeto de trabalho de conclusão de curso) e relatório (portfólio e relatório de estágio) possuem de 10 a 30 páginas
- Os trabalhos de grande porte, como o Trabalho de Conclusão de Curso (TCC), a dissertação e a tese têm mais de 30 páginas.
Formatação nas Normas da ABNT
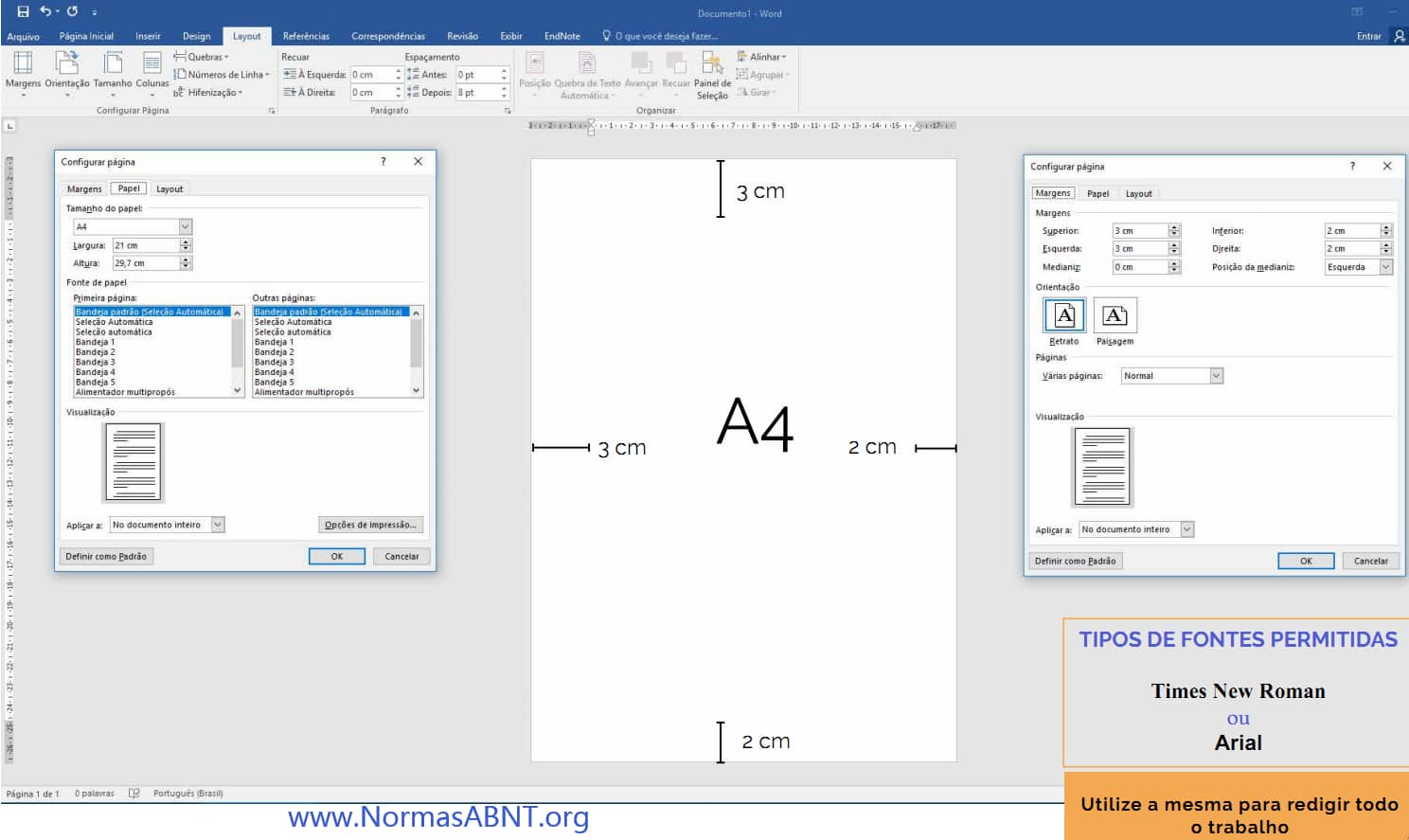
Esse é um critério importante para desenvolver bem o trabalho e ele estar completamente de acordo com o que a norma solicita de quem desenvolve seu trabalho de conclusão.
O primeiro ponto a ser frisado é as margens essa deverá ter no papel A4 impresso o texto na margem superior e esquerda com 3cm e a margem inferior e direita deve ser 2cm. (Clique aqui para ver como configurar o layout, margens)
Nada de imagens ao fundo com marca d´agua e papel em branco.
Outro aspecto que deve conter é a paginação, essa só começa a partir da introdução, como também é importante deixa o texto sempre com uma fonte e tamanho padrão, essa seria na fonte times new roman ou arial, em um tamanho padrão 12. sendo esse de espaçamento 1,5cm entre linhas e recuo como também deverá ter uma média de 1,25 cm no início de cada parágrafo.
As citações no texto mudam, esse deve ser entendido que se tiver citação com até 3 linhas, essa não precisará de formatação especial. Para as citações com 4 linhas ou mais que isso essa deverá ser em um recuo de 4 ou 6 cm na margem esquerda, com sua fonte 10.
Essas são as regras básicas que um trabalho, monografia deve ter, claro que, essas regras contêm mais complexidade, porém o estudante pode se aprofundar ainda mais para que seu projeto fique o mais completo possível.
Tamanho de fonte e espaçamento
Veja a tabela seguinte sobre tamanhos de fonte
| Tamanho de fonte | Onde usar |
|---|---|
| 12 | Pré-textual: capa, folha de rosto, ficha catalográfica, dedicatória, agradecimento, epigrafe, resumo e abstract, lista de ilustrações, tabelas, abreviaturas e siglas, símbolos, sumário Textual: Introdução, desenvolvimento, conclusão Pós-textual: referências, glossário, apêndices e anexos |
| 10 | Textual: citações textuais entre aspas de mais de três linhas, paginação, notas de rodapé, legendas, texto da fonte das ilustrações, figuras e tabelas |
Conforme ABNT NBR 14724:2011 atualizada. (baixe de grátis em PDF)
veja a tabela seguinte sobre os espaçamentos ABNT
| Espaçamento | Onde usar |
|---|---|
| 1,5 | pré-texto: capa, folha de rosto, dedicatória, agradecimentos, epigrafe, listas e sumário textual: introdução, desenvolvimento e conclusão pós-texto: glossário, apêndice e anexo |
| simples | pré-texto: resumo e abstract textual: citações de mais de três linhas, notas de rodapé, legendas e fontes de ilustrações, paginação pós-texto: referências |
Leia os artigos relevantes
- Formatação ABNT – Capa, parágrafo, sumário, citação, referências
- Formatação TCC e Monografia (Resumo e exemplos)
Tipos de citações
💡 Citação direta curta e exemplos
💡 Citação direta longa e exemplos
A Norma ABNT NBR 10520 que especifica as características para apresentação de citações é atualizada em agosto de 2023. A versão mais recente é NBR 10520/2023. Os dois principais mudanças em 2023 são:
A autoria (pessoa física ou jurídica) e títulos devem figurar em letras maiúsculas e minúsculas. (antes era CAIXA ALTA)
O ponto final deve ser inserido somente ao final da sentença!
Citação direta
É uma cópia literal do texto. Transcrevem-se geralmente decretos, regulamentos, leis, fórmulas científicas ou trechos de obras. O tamanho da citação determinará sua localização no trabalho.
Se a citação tiver até três linhas, virá incorporada ao parágrafo, entre aspas duplas. As citações com mais de três linhas ficarão abaixo do parágrafo, em bloco, com início sob a linha anterior, a quatro cm à direita da tabulação, em espaço simples.
Exemplo de citação direta curta:

Exemplo de citação direta longa:

Citação indireta
Nas norma da ABNT não existe diferenciação entre citação indireta longa e curta. Além disso, na citação indireta não há (nem tem como haver, na prática) necessidade de recuo em relação à margem esquerda, nem de redução do tamanho da fonte. Tal citação deve estar incorporada, de forma fluída, no corpo do texto. Revejam suas orientações! Não pode conter aspas e é opcional incluir o número de página e localização.
Exemplo de citação indireta:
De acordo com Silva (2021), a tecnologia está rapidamente transformando nossa forma de viver.
Citação de citação
É a menção de um documento ao qual não se teve acesso diretamente. Ocorre quando o pesquisador encontra uma citação dentro de uma obra que está consultando.
Exemplo 1

Exemplo 2

Leia como usar Citações.
Modelos prontos ABNT
Veja os modelos prontos nas regras ABNT, e se tiver interesse nos modelos prontos completos, pode pagar R$10 e receberá três arquivos (MS Word) de modelo de trabalho pronto ABNT no seu E-mail ( Clique Aqui para Pagar). E meramente pelo objetivo educacional, compartilhamos os modelos prontos em Word, para as áreas como:
- Administração
- Design
- Engenharia
- Direito
- Odontologia
- Ciências Biológicas
- Economia
- Medicina, etc
Modelo CAPA ABNT

Modelo Folha de Rosto ABNT

Modelo Sumário ABNT

Referências Bibliográficas
Se quiser aprender como fazer referências bibliográficas pela ABNT, veja os exemplos de referências dos livros, blogs, páginas da internet, twitter, youtube, wikepedia etc, clique no link:
Leia também







